コロナウイルスには、パンデミック(世界的大流行)を引き起こしている新型コロナウイルス(SARS-CoV-2)など、さまざまな種類がある。これらのうちSARS(重症急性呼吸器症候群)関連コロナウイルス同士のゲノム(全遺伝情報)を比べたところ、特定の領域に未知の仕組みによる可能性のある、長い塩基配列の挿入や欠失、入れ替わりが集中して起きていることが分かった。東北大学の研究グループが発表した。さらなる変異への対応や対策を考える手がかりとなる可能性がある。
コロナウイルスには風邪の病原体となるタイプや、重篤な肺炎などを引き起こすタイプがある。後者には2003年に見つかり大流行したSARSコロナウイルス(SARS-CoV)や、2012年に見つかったMERS(中東呼吸器症候群)コロナウイルス(MERS-CoV)などがある。新型コロナウイルスは、SARSコロナウイルスの仲間に分類されている。
コロナウイルスやインフルエンザウイルスは、人と動物に共通する感染症。人にパンデミックを引き起こす段階のゲノム配列は、その前に別の動物に宿った段階のものと比べ、どんな領域にどんな変異が起きているのだろうか。さまざまな動物から採取されたウイルスのゲノム配列を比べることは、今後の変異や進化で新たに起こる感染への対策のために重要だ。
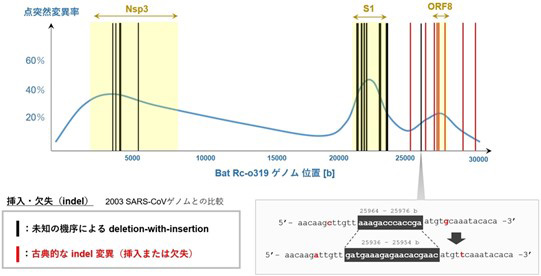
そこで研究グループは、2003年に中国で採取されたSARS-CoVと、13年に東京大学のグループが岩手県の洞窟のコウモリから採取したSARS関連コロナウイルス(Rc-o319)のゲノムを比べ、ウイルスが進化の過程でどう変異するのかを探った。両者は配列がよく似ており、共通の祖先がいたと思われる。
その結果、Rc-o319のゲノムには、SARS-CoVに比べ少なくとも30カ所以上の挿入や欠失があり、その多くが10塩基以上、所により100塩基をも超える比較的長い配列に及んでいた。また、その半数近くで挿入と欠失が同時に起き、一定の長さの配列が“ゴソッと”入れ替わるタイプの変異が頻繁にみられた。これらは1塩基が入れ替わったり、少数が挿入されたり欠失したりする古典的に知られる変異とは明らかに異なる。しかもゲノムの2つの領域に集中していることから、単なる偶然ではなく、何らかの未知の仕組みによる可能性がある。

Rc-o319ゲノムに挿入された長い配列の起源を探ろうと、それらの全てをSARS-CoVとRc-o319のゲノム中に探したが、似たものはなかった。こうしたことから研究チームは、挿入された配列はウイルス自身ではなく、宿主である動物の細胞や別の微生物など、他者の配列に由来するとみられると判断した。また、Rc-o319ゲノムの挿入や欠失の大半は、SARS-CoVと2019年のSARS-CoV-2の間に見られる挿入や欠失にはなかった。日本のコウモリなど、宿主の体内で生じたと考えられる。
SARS関連コロナウイルスでは、こうした比較的長い挿入や欠失が特定のゲノム領域に蓄積し、変異の重要な要素となった可能性がある。今後、こうした変異を引き起こす宿主の要因や未知の仕組みを解明できれば、同ウイルスの変異で起こる問題への対処に役立つ期待があるという。
研究グループの東北大学大学院医学系研究科の赤石哲也助教(漢方・統合医療学)は「10塩基以上の挿入と欠失が、偶然とは思えないほど同じ領域に高頻度に起こっていたのは意外なことだった。この現象の仕組みや、挿入された塩基の由来、そして人間に起こるパンデミックへの関与度など、非常に興味深い」と述べている。
成果は米微生物学会の学術誌「ジャーナル・オブ・ビロロジー」の電子版に7月21日に掲載され、東北大学が25日に発表した。
関連リンク
- 東北大学プレスリリース「コロナウイルスのゲノムに蓄積した進化の痕跡を発見 コロナウイルスがもたらすパンデミックの機序解明へ期待」




