ヒトゲノムの解読は、2003年に完了し、全配列が決定した。決定したデータは国際参照配列※1として公開され、今では世界中のゲノム研究を支えている。だが今年4月、東北大学東北メディカル・メガバンク機構は、この国際参照配列の日本人版とも言える「日本人の基準ゲノム配列」を作成した。国際参照配列との違いは何だろうか。それを可能にした高度な技術や、もたらす利益についても紹介する。
※1 国際(ヒトゲノム)参照配列/ヒトゲノムの全染色体の塩基配列。学術組織ゲノムリファレンスコンソーシアムが管理し、繰り返し改訂を行っている。今最も新しいのは「GRCh38v1」というバージョン。
日本人特有のゲノム配列がある?
ゲノムは生命の設計図とも言われ、その本体はDNAという長いひものような物質だ。DNAは、4種類のヌクレオチドという化学物質の長いつながりで、それぞれ、A(アデニン)、G(グアニン)、T(チミン)、C(シトシン)と示す。このAGTCの数(塩基数)や並び方(塩基配列)が、あらゆる生命の営みを決めている。
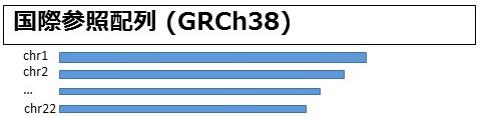
ヒトゲノムの塩基数は30億である。冒頭で述べたようにその塩基配列は国際参照配列として公開されており、今日のゲノム研究に欠かせない。しかし、基になっているゲノムは主に欧米人のものだ。もし日本人特有のゲノム配列があったとしたら、それは国際参照配列には含まれていない可能性がある。
そこで、東北大学東北メディカル・メガバンク機構は、日本人のゲノムを解析し、国際参照配列に含まれない新しい塩基配列を見出した。塩基数にして合計約200万以上にのぼる。これを、国際参照配列内の該当する3,000箇所以上に挿入し、日本人基準ゲノム「JRGv1」(Japanese Reference Genome version 1)と名付けた。さらにv2、v3と改善していく必要があるが、「第一歩として見つけられた数には満足している」と研究チームは話している。
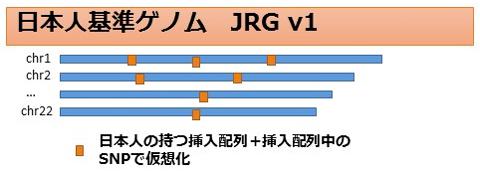
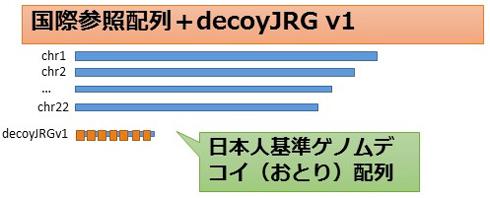
場合によっては、国際参照配列をそのまま残した方が研究者に活用されやすい。新たに見つけた配列のみをまとめたデータをデコイ(おとり)配列「decoyJRGv1」と名付け、日本人基準ゲノムと併せて公開する予定であるとした。
最新鋭のゲノム解析技術を駆使して
今回の成果を支えたのが、30億も連なるDNAの塩基配列を読み取る「DNAシークエンシング」や、読み取った情報を処理する「バイオインフォマティクス」といった技術だ。現在の技術では、30億の塩基配列を切らずに読むことはできないので、DNAを適度な長さに切り、次世代シークエンサー※2にかける。本プロジェクトでは、一度に1万塩基以上解析できる長鎖読み取り型次世代シークエンサーで、平均1.2万塩基に切ったDNA断片を一気に読み取った。このシークエンサーは従来の短鎖読み取り型に比べてエラーが多いが、ゲノム一人分あたり100回以上繰り返すことで、精度を上げた。
※2 次世代シークエンサー/ランダムに切ったDNAの塩基配列を読み取る機器。一度に数百塩基を読み取る短鎖読み取り型と、数千以上読み取る長鎖読み取り型がある。一般のゲノム研究では、速くて精度も高い短鎖読み取り型が使われる。

読み取った断片情報は、「デノボアセンブル」と呼ばれる情報科学の手法でつなぎ合わせる。デノボアセンブルは、隣接する断片情報間の重複する部分を手掛かりに、未知の塩基配列を決定する方法だ。ヒトのゲノム情報を100回も繰り返し読み取った大量のデータを処理するのは、スーパーコンピュータを使っても数カ月かかる大作業だ。
ゲノム研究を支える基準ゲノム
なぜ日本人の基準ゲノムが必要なのだろうか?例えば、研究者が日本人のゲノム情報を基にある病気へのかかりやすさを調べる場合、病気にかかりやすい遺伝子にたどり着くためには「ゲノムを読み取る」ステップが必要だ。
とはいっても、先に紹介したようにゼロから読み取るわけではない。DNAを短く切って、次世代シークエンサーで読み、バラバラになったDNAを、「お手本」に習って元の配列に復元し、「読み取る」のだ。この「お手本」となるのが基準ゲノムである。
もしお手本が間違っていれば、ゲノムを元通りにできず、読み取りの精度が下がってしまう。日本人のゲノムなら、日本人の基準ゲノムをお手本にすればより精確に読み取ることができ、注目する領域についてもより精確に調べられる。研究チームは、今回の成果を、日本の医学研究の大きな基盤となるだろうと期待している。
東北から広がる医療の可能性
本研究で解析に使われたゲノムは、東北地方に住む成人から提供された。その提供者たちは、東日本大震災の復興事業として2012年に始まった「東北メディカル・メガバンク計画」のコホート調査※3の参加者だ。
東北メディカル・メガバンク計画は、東北地方の人びとの生体試料や医療・ゲノム情報のバイオバンクで、10年計画のコホート調査で15万人の登録を目指している。本プロジェクトは日本の三大バイオバンクの一つとされ、病気の人ではなく一般住民が対象であることや、世界中の研究者が集積した生体試料やゲノム情報などのデータを使えるのが特徴だ。15万人規模のバイオバンクは世界でも比較的大きい方で、特に、本プロジェクトで実施されている「三世代コホート」は、同様のものに成功例が少ないこともあり注目されている。
※3 コホート調査/生活習慣や環境が、人の健康状態にどのような影響を与えるのかを知るために、特定の地域や集団に属する人を一定期間(数年?数十年)観察する調査。多大な時間とコストがかかるが、因果関係が最も明確に分かる疫学的手法。
今回の基準ゲノム決定に先立って、1070人分の全ゲノム解析データをもとに構築した、全ゲノムリファレンスパネルの情報が公開されている(下の図参照)。
このデータから、既に2,000万個以上の一塩基多様体※4が見つかっている。これらを詳しく調べれば、病気のリスクや薬への反応などの個人差について分かると言われている。例えば、かかりやすい病気が分かれば生活習慣を改善して未然に防げるし、薬物代謝に関わる遺伝子を調べて薬の効き方が予測できれば、効果を高め、副作用を最小限にする投薬が可能になるのだ。
※4 一塩基多様体/集団内で、ゲノム配列中の塩基がひとつ違っている箇所で、個人間の違いの原因と考えられている。
プロジェクトのこれから
個人のゲノム配列の読み取りから日本人の基準ゲノムの決定を遂げ、次はどこに向かっていくのだろうか。プロジェクトの今後について研究チームに伺った。
Q. 本プロジェクトの次のステップは何ですか?
A. 今回の基準ゲノム作成では、日本人ゲノムと国際参照配列との比較を行い、国際参照配列には存在しない新規な配列の検出と解析を行いました。次は別の種類の変異についての解析を行います。例えば国際参照配列にあって日本人ゲノムにはない配列や、長いゲノム配列の一部分のみ向きが逆転している変異などです。こうした変異の解析結果を加えていくことで、より精緻で有用な基準ゲノムを作成することを目指しています。
Q. それを阻む要素として、どんなことが考えられますか?
A. やはり、多数の日本人の方のDNAのシークエンスを、長鎖読み取り方式で行う必要があります。日本人を100人読むことで、100人に1人が持つぐらいの違いを検出できると考えています。本当は、1,000人規模で読めれば、かなり緻密な情報が得られると思います。ただ、長鎖読み取り方式は結構な費用がかかるので、現状では世界中あらゆる人種の情報をかき集めても、長鎖読み取り方式で読まれたDNA情報は100人分にもならない状況です。
Q. メディカルメガバンクを構築することで、最終的に目指すことを教えてください。
A. 個別化予防、医療を日本で実現するための基盤づくりとその実現が大目標になります。そのためには、まず、個人の個性を理解する必要があります。その情報のひとつがゲノム情報です。もちろん、ゲノム情報だけで個性は決まりませんが、重要な要素のひとつであることは変わりません。
残念ながら、今まで日本には、全ゲノムの詳細な情報はありませんでした。今回の日本人の基準ゲノムの作成は、その基盤情報の整備活動のひとつになります。今後は、これらのゲノム情報を拡充するとともに、日本のコミュニティでこれらの基盤情報を活用してもらい、大目標を一緒に実現していきたいと考えています。
忘れてはならないゲノム研究の倫理的側面
特定の人種や民族に特徴的な遺伝子やゲノムの研究は、それぞれに適したより良い医療の実現に貢献する一方で、倫理、社会、法的な問題(ELSI。Ethical, Legal, and Social Implications)を生む可能性もある。世界には、複数の民族や人種のDNAを調べるプロジェクトもあるが、特定の民族や人種にレッテルを貼るような結果や、遺伝的アイデンティティと社会的アイデンティティのギャップを生むような結果をもたらしてはいけない。また、究極の個人情報であるゲノム情報がひとり歩きして、個人に不利益となるリスクはないだろうか。
研究チームは、未来の技術をすべて予測することは困難だが、公開された情報のみから個人が特定されないように、最大限の配慮を行うべきだと考えている。そのため、ゲノム情報を公開する際には、個人が特定され、特定の人々に対する差別につながらないよう、倫理審査機関はもとより、ゲノム解析の専門家とも慎重な検討を行っていると話す。
ゲノム科学の発展に期待しつつも、これが抱える問題も見据えていきたい。
(サイエンスライター 丸山 恵)





